发布时间:2025-05-28 14: 17: 00
在分子生物学实验设计中,序列数据的规范管理和项目的安全备份,是确保科研流程顺利进行的重要保障。尤其是在使用功能强大的Oligo软件进行引物设计、寡核苷酸分析、结构预测等工作时,若不做好数据管理与备份,一旦发生系统崩溃或数据丢失,后果将极为严重。本文将详细讲解Oligo软件如何管理序列数据 Oligo软件如何备份项目数据,帮助用户建立起稳定、高效、安全的科研数据处理流程。
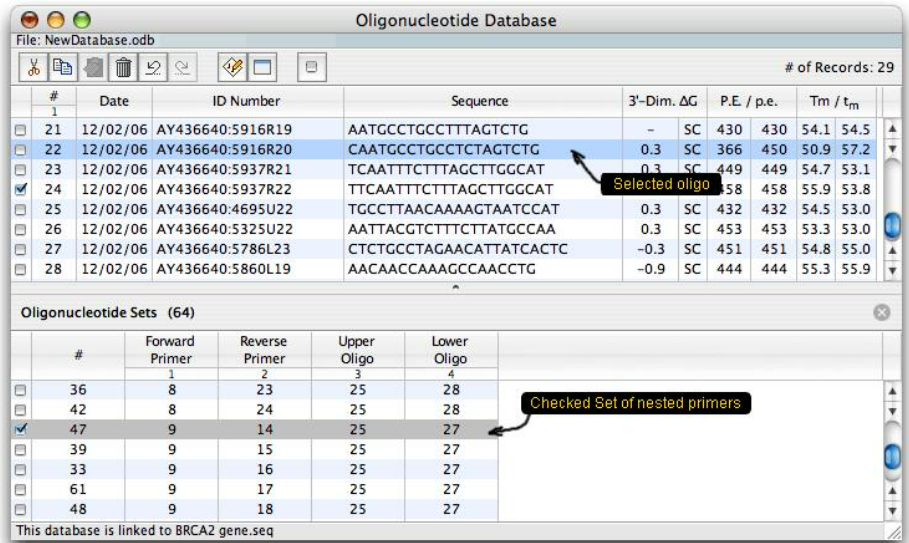
一、Oligo软件如何管理序列数据
Oligo作为一款以寡核苷酸设计与分析为核心的工具,提供了完整的序列数据管理方案。你不仅可以导入各种格式的核酸序列,还能对其进行分类、命名、注释、保存与检索。
1. 支持的序列格式与导入方式
Oligo支持多种常见的核酸序列输入格式,包括但不限于:
纯文本(.txt):直接粘贴或载入碱基序列;
FASTA格式(.fasta / .fa):支持带有注释行的标准DNA序列;
GenBank格式(.gb / .gbk):支持结构化注释,如CDS、启动子、ORF等。
导入操作步骤如下:
打开Oligo主程序 → 点击“File → Open Sequence”;
选择你准备好的序列文件,点击打开;
程序会自动识别序列并加载至当前工作界面;
可通过“Sequence → Properties”自定义序列名称、类型(线性/环状)、起止位点等信息。
2. 序列分组与命名建议
每个序列文件建议以样本名 + 目标区域 + 时间命名,如“p53_exon4_Jan24.fas”;
使用“Save As”功能按实验项目分类保存,如“Project_CancerGene/TP53/Design1.oligo”;
可在序列窗口中使用“Notes”功能添加功能性描述,如“contains TATA box”、“mutant at 168G→A”等;
对引物设计区域使用颜色标注,有助于后期审查与汇报。
3. 多序列项目组织策略
虽然Oligo不像SnapGene那样具备多标签项目管理界面,但你可以采用以下策略组织多个序列:
使用项目文件夹方式管理:将同一实验项目的所有序列文件存于同一目录;
按目标基因/片段编号建立子文件夹,如“GAPDH”、“Beta-Actin”、“Vector-pCDNA3.1”;
每个序列建议搭配说明文档(如.txt、.docx)记录序列来源、用途与设计人,便于多人协作。
4. 快速检索与版本管理建议
为每个重要序列文件保留多个版本,如“Design_v1.oligo”、“Design_v2_mut.oligo”,避免误操作覆盖;
利用Windows文件资源管理器的“最近修改时间”排序可快速定位最新文件;
将核心数据汇总为一个“README.txt”文档,记录各版本序列及对应实验日期与用途。
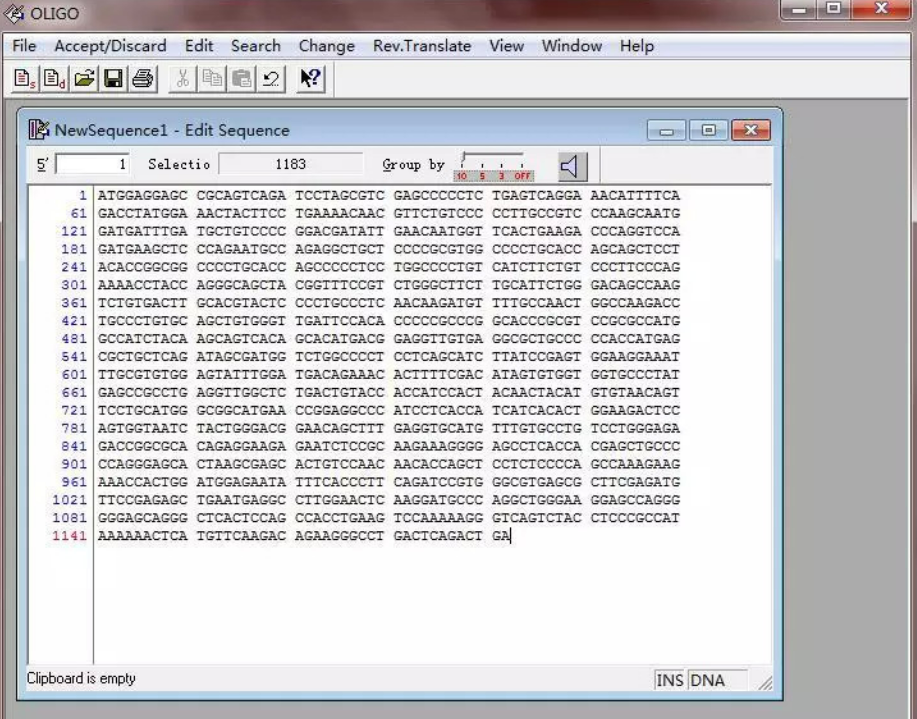
二、Oligo软件如何备份项目数据
序列数据的设计与分析往往需要长时间投入,若不定期备份,一旦数据丢失将会影响整个实验进度。以下是Oligo项目数据的完整备份方法与策略。
1. 理解Oligo文件结构
Oligo软件的数据主要由以下文件构成:
.oligo 文件:Oligo的原生项目文件,保存序列、引物设计、注释、结构分析等所有内容;
.txt/.fas/.gb 文件:导入或导出的序列数据文件;
Export文件(如.csv/.pdf/.doc):导出的分析结果、报告图表、引物参数表等。
这些文件构成了完整的设计数据包,建议统一打包归档备份。
2. 手动备份操作流程
步骤一:整理文件夹
在项目主目录下建立如“Backup_2025_05_23”文件夹;
将当前所有“.oligo”、“.fasta”、“.txt”及分析结果复制至该文件夹中。
步骤二:创建压缩档案
使用WinRAR、7-Zip等工具将文件夹压缩为“Project_X_Backup_2025_05_23.zip”;
文件命名中包含日期,有利于追踪版本变化。
步骤三:多渠道备份
本地硬盘(如D盘)、移动U盘、外接移动硬盘;
云端同步服务,如 OneDrive、Google Drive、百度网盘、阿里云盘;
重要项目建议上传至实验室服务器或NAS系统。
3. 自动化备份建议
虽然Oligo本身不具备自动备份功能,但你可以借助以下策略:
使用 Windows 定时任务,每天自动备份项目文件夹;
安装如 FreeFileSync、SyncBackFree 等工具,设定“每天18:00同步至备份盘”;
Git版本管理:将Oligo项目导出的序列(.txt/.fa)版本化管理,跟踪变更轨迹。
4. 备份前注意事项
确保所有文件已关闭再压缩,避免损坏;
清理中间无用文件,减少空间占用;
使用统一格式命名文件夹与备份包,便于长期归档检索。
5. 误删或崩溃后的数据恢复策略
若不慎关闭未保存的文件,检查Oligo是否生成缓存文件(部分版本支持恢复上次编辑状态);
使用数据恢复软件(如Recuva、EaseUS)尝试找回误删的.oligo项目文件;
若为Git同步目录,可检出历史版本恢复文件。
三、如何在团队合作中建立标准化的Oligo数据流转机制
在实验室或项目组层面,Oligo不只是一个单人使用的工具,越来越多的团队希望实现多人协作、跨机器工作、分工明确的设计流程。这时候,就需要一个明确的Oligo数据流转机制,来保障设计、校对、汇总、备份等环节的稳定运行。
1. 建立统一的文件命名与目录规范
所有成员按照项目编号/任务类型/日期的方式统一命名文件,例如:
Project_GeneX/PromoterDesign/TP53_Design_2025_0523.oligo
每位成员负责的文件需在命名前标注姓名或缩写,便于追溯和责任划分。
2. 设置共享存储空间
使用NAS服务器、云盘(如Google Drive、OneDrive)、或内部共享网盘作为主项目目录;
所有成员的Oligo序列文件、引物报告、导出文件统一上传至该目录;
可设置“只读/可编辑”权限,保障数据不被误删。
3. 制定版本控制机制
每轮修改后在文件名加上版本号,如v1、v2_final、v3_review;
使用Excel建立“版本登记表”,记录每个版本的修改内容、修改人、修改时间;
关键文件每月统一备份一次,形成“月度归档”。
4. 配套数据说明文档
所有设计项目需配套一个README.txt,记录项目背景、序列来源、设计目标、使用的参数、注意事项等;
尤其在涉及多个相似设计方案(如启动子变体、引物组组合)时,文档说明能极大减少沟通成本。
5. 指定数据管理员定期巡查
实验室或团队指定一位成员定期检查Oligo项目文件夹的完整性;
确保每个成员提交数据后有备份、有版本记录;
一旦发现重复命名、缺失数据、无说明文件,及时纠正。
通过这一系列的机制,不仅能让Oligo的数据使用更专业、更系统,也能帮助团队在面对项目复现、审稿答辩、人员更替时,拥有更强的组织力与透明度。
总结
本文围绕“Oligo软件如何管理序列数据 Oligo软件如何备份项目数据”两个主题,从文件格式支持、序列组织规范、项目版本控制,到多维度备份策略进行了系统讲解。通过良好的数据管理机制和定期备份习惯,用户可以有效保障研究数据的完整性与安全性。
在分子生物学研究逐渐走向自动化与大规模数据处理的今天,掌握这些“看似基础”的数据管理技能,将在关键时刻为你的项目保驾护航。
展开阅读全文
︾
读者也喜欢这些内容:
反转录过程中常用的引物有哪几类 反转录用随机引物还是olig
在RNA研究与转录组分析中,反转录是基础又关键的一步,它将RNA模板转录成cDNA,为后续PCR扩增、qPCR定量、RNA测序等实验打下基础。实验结果的准确性与反转录使用的引物类型有密切关系。因此,“反转录过程中常用的引物有哪几类,反转录用随机引物还是oligo”成为科研工作者特别关注的问题。本文将围绕这两个核心主题展开详细说明,以提升实验效率与数据质量。...
阅读全文 >
Oligo引物和random引物区别 Oligo dt和随机引物作用一样吗
在分子生物学实验中,逆转录是将RNA转录为cDNA的重要步骤,而引物的选择直接决定了逆转录的效率和特异性。“Oligo引物和random引物区别,Oligodt和随机引物作用一样吗”是实验新手和科研人员经常关注的问题。无论是在qPCR、RNA测序还是文库构建前期,引物选择都会影响到后续分析的可靠性和数据一致性。为了更好地理解这两个引物类型的差异及其在实验中的应用效果,本文将通过三个层面详细分析,并补充扩展一类常见但易被忽视的引物——基因特异性引物。...
阅读全文 >
引物设计的原则有哪些 如何用Oligo软件设计引物
引物设计在分子生物学实验中至关重要,尤其在PCR扩增、测序、突变分析等场景中,一对高质量的引物往往决定整个实验是否成功。面对复杂的基因组信息与实验需求,科学合理地设计引物成为每个科研人员必备的技能。Oligo软件作为专业的引物设计工具,因其高精度、参数可控、结构分析功能强大,在科研工作中被广泛应用。本文围绕“引物设计的原则有哪些,如何用Oligo软件设计引物”这一主题,深入讲解引物设计的核心原则、Oligo操作方法,帮助科研人员提高实验效率和数据可靠性。...
阅读全文 >
Oligo软件如何统计序列信息 Oligo怎么导出统计结果
在分子生物学的研究与实验中,对序列的统计分析是一项非常基础却关键的工作。无论是GC含量、碱基组成,还是序列长度与熔解温度(Tm),这些统计信息都直接影响到引物设计、克隆构建、合成策略等各个方面。Oligo软件如何统计序列信息Oligo怎么导出统计结果,成为许多科研人员在实际操作中经常遇到的需求。本文将围绕这个主题,详细介绍Oligo软件中与序列统计相关的核心功能、使用方法和结果导出技巧,帮助用户更高效地获取并利用这些关键数据。...
阅读全文 >